সুচিপত্র:
- লেখক John Day [email protected].
- Public 2024-01-30 08:01.
- সর্বশেষ পরিবর্তিত 2025-01-23 14:36.
লিখেছেন: মধুমিতা কানন, হেনরি এনগুয়েন, অ্যাশলে উরুতিয়া আভিলা, মেই জিন
এই ম্যাটল্যাব কোডটি একটি রোগীর মস্তিষ্কের এমআরআই স্ক্যানের মধ্যে পাওয়া টিউমারের সঠিক আকার, আকৃতি এবং অবস্থান সনাক্ত করার জন্য একটি প্রোগ্রাম। এই প্রোগ্রামটি মূলত মস্তিষ্কের এমআরআই স্ক্যানগুলিতে টিউমার সনাক্তকরণের সাথে কাজ করার জন্য ডিজাইন করা হয়েছে, তবে এটি অন্যান্য অঙ্গ স্ক্যানগুলিতেও ক্যান্সার নির্ণয়ের জন্য ব্যবহার করা যেতে পারে।
নিম্নলিখিত নির্দেশাবলী প্রথমে ফিল্টারিং এবং এমআরআই স্ক্যান পরিষ্কারের মাধ্যমে চিত্র বিশ্লেষণের পদ্ধতিগুলি বর্ণনা করবে, বাইনারাইজিং, মিডিয়ান ফিল্টারিং এবং স্লাইডিং উইন্ডোর মাধ্যমে। এরপরে, এটি একটি পূর্ব-উত্পন্ন উপবৃত্তাকার মুখোশ ব্যবহার করে কীভাবে টিউমারকে বিচ্ছিন্ন করতে হবে এবং টিউমারের আকারের পরিধি রূপরেখা করার জন্য এটি আরও ফিল্টার করার নির্দেশ দেবে।
একবার টিউমার ধরা পড়লে নির্দেশাবলী আরও বর্ণনা করবে কিভাবে এই প্রোগ্রামটিকে গ্রাফিক্যাল ইউজার ইন্টারফেসে (GUI) অন্তর্ভুক্ত করা যায়। এই নির্দেশাবলী জুড়ে, এই এমআরআই স্ক্যান বিশ্লেষণ কিভাবে কাজ করে তা ব্যাখ্যা করতে উপযুক্ত কোড এবং ফাইল সংযুক্ত করা হবে।
এই নির্দেশের সাথে এগিয়ে যাওয়ার আগে কিছু জিনিস জানতে, ডাউনলোড করতে এবং প্রস্তুত করতে হবে: 1। MATLAB এর সর্বশেষ সংস্করণটি ডাউনলোড করা নিশ্চিত করুন। আপনি এখানে R2018b ইনস্টল করতে পারেন:
2. এই প্রোগ্রামটি চালানোর জন্য, আপনার এমআরআই মস্তিষ্ক স্ক্যান ফাইলগুলিতে অ্যাক্সেস থাকতে হবে। যদিও কিছু সবসময় গুগল ইমেজ থেকে পাওয়া যায়, প্রতিটি রোগীর মস্তিষ্ক স্ক্যানের বিভিন্ন স্তরের সঠিক ছবি থেকে পুঙ্খানুপুঙ্খ এবং সঠিক বিশ্লেষণ করা যেতে পারে। আপনি এই ডাটাবেস থেকে গ্লিওব্লাস্টোমা প্রি এবং পোস্ট ট্রিটমেন্টের 20 টি ভিন্ন রোগীর ফাইল অ্যাক্সেস করতে পারেন:
3. এই কর্মসূচির ফোকাস এবং বিভিন্ন প্রকল্প যা এই প্রকল্পকে নির্দেশ করে এই গবেষণাপত্রে বর্ণিত হয়েছে:
ধাপ 1: গ্রাফিকাল ইউজার ইন্টারফেস (GUI) শুরু করুন
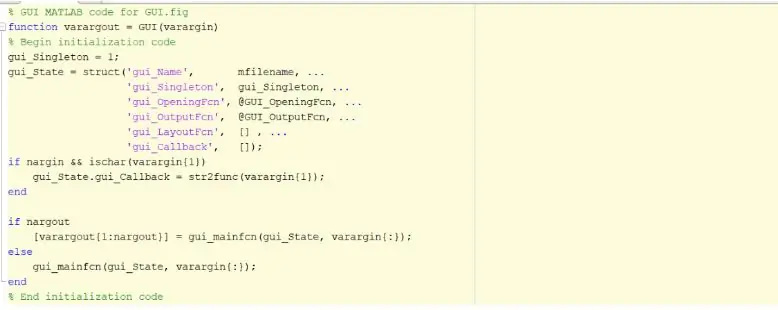
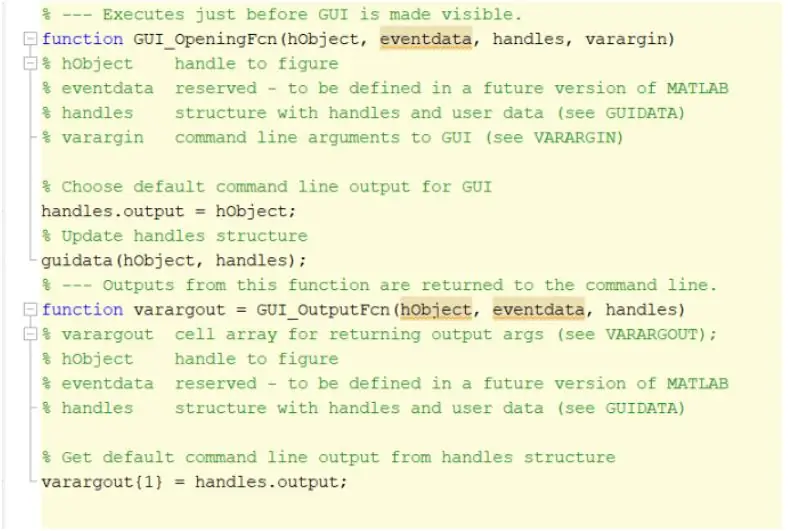
প্রথম ধাপটি হবে গ্রাফিক্যাল ইউজার ইন্টারফেস, GUI তৈরি এবং আরম্ভ করা। কমান্ড উইন্ডোতে গাইড টাইপ করে, এন্টার টিপে এবং একটি নতুন জিইউআই তৈরি করে এটি করা যেতে পারে। একবার এই ধাপটি সম্পন্ন হলে আপনি অক্ষ, স্থির পাঠ্য, সম্পাদনা পাঠ্য এবং পুশ বোতামগুলির মতো ফাংশন তৈরি করতে শুরু করতে পারেন যা প্রোগ্রামটি চালানোর পরে প্রদর্শিত হবে এবং ব্যবহারকারীর সাথে যোগাযোগ করতে পারে। এই ফাংশনগুলি সম্পদ পরিদর্শকের মাধ্যমে সম্পাদনা এবং ম্যানিপুলেট করা যেতে পারে, তবে এই ফাংশনগুলি তৈরি করার সময় সবচেয়ে গুরুত্বপূর্ণ বৈশিষ্ট্যটি পরিবর্তন করতে হবে তা হল ট্যাগ নাম। বাস্তবায়িত প্রতিটি ফাংশনের ট্যাগ নাম পরিবর্তন করা গুরুত্বপূর্ণ কারণ এটি আমাদের একটি আলাদা কলব্যাক ফাংশন তৈরি করতে দেবে। একবার আপনি আপনার GUI এর বিন্যাসে সন্তুষ্ট হয়ে গেলে আপনি DICOM ফাইলগুলি লোড করতে এগিয়ে যেতে পারেন যা GUI এর মধ্যে প্রদর্শিত হবে।
ধাপ 2: MATLAB এ MRI ছবি লোড করা এবং পড়া
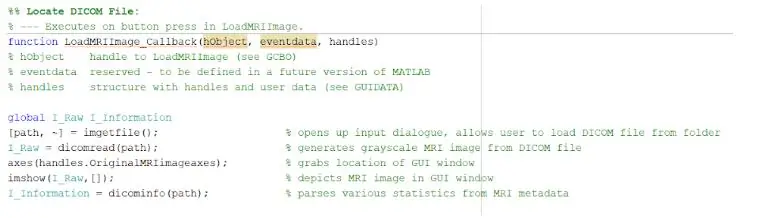
DICOM ফাইলগুলি লোড করার জন্য আপনাকে কল ব্যাক ফাংশনটি সঠিকভাবে শুরু করতে হবে যা "লোড এমআরআই ইমেজ" বোতাম টিপে কার্যকর করা হবে। একবার এটি সম্পন্ন হলে, আপনাকে অবশ্যই একটি গ্লোবাল ভেরিয়েবল তৈরি করতে হবে যা হ্যান্ডেল অক্ষগুলিতে চিত্রটি প্রদর্শন করবে যেখানে আপনি মূল এমআরআই চিত্রটি প্রদর্শন করতে চান। ডাটাবেস থেকে ডাউনলোড করা এমআরআই স্ক্যান ইমেজগুলি হল সব DICOM ফরম্যাট করা ফাইল যা আপনার MATLAB ডিরেক্টরিতে লোড করা প্রয়োজন। প্রোগ্রামটিতে লোড করার জন্য imgetfile ব্যবহার করে ফাইলটি সনাক্ত করুন। অন্তর্নির্মিত ম্যাটল্যাব ফাংশন 'ডিকোম্রেড' ব্যবহার করে ছবিগুলি পড়া হয় এবং প্রতিটি ফাইলের জন্য প্রথম কাঁচা ছবিটি ইমশো ব্যবহার করে বাম জিইউআই অক্ষের মধ্যে সংযুক্ত করা হয়।
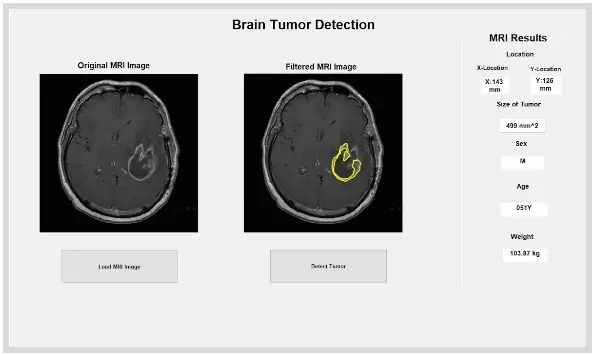
MATLAB ফাংশন 'ডিকোমিনফো' প্রতিটি এমআরআই ডিকম ফাইলের সমস্ত তথ্য সম্বন্ধে অত্যন্ত কার্যকর। আমরা রোগীদের সমস্ত বর্ণনামূলক তথ্য যেমন তাদের লিঙ্গ, বয়স, ওজন এবং উচ্চতা বের করতে এই ফাংশনটি ব্যবহার করেছি। এই ফাংশনটি আপনাকে স্ট্যাক অর্ডারও প্রদান করে যা গ্রাফিক্যাল ইউজার ইন্টারফেসের মধ্যে প্রোগ্রামটি বাস্তবায়নের জন্য দরকারী। আমরা রোগীদের প্রতিটি বর্ণনামূলক তথ্যের জন্য ভেরিয়েবল তৈরি করেছি যা সনাক্তকরণ বোতাম টিপলে GUI- এর জন্য ব্যবহার করা হবে।
ধাপ 3: চিত্র ফিল্টারিং

একবার কাঁচা ছবির DICOM ফাইল লোড হয়ে পড়ার পরে, ছবিটি গ্রেস্কেল থেকে শুধুমাত্র কালো এবং সাদা পিক্সেলের সমন্বয়ে একটি বাইনারাইজড রূপে রূপান্তরিত করতে হবে আমরা কাঁচা ছবি থেকে একটি বাইনারি ইমেজ তৈরি করতে 'imbinarize' ফাংশনটি ব্যবহার করেছি 0.59 সংবেদনশীলতা মান অভিযোজিত থ্রেশহোল্ডিং দিক নিয়ন্ত্রণ ডিফল্ট থ্রেশহোল্ড সংবেদনশীলতা ফ্যাক্টর, 0.5 কম ছিল এবং ছবি থেকে উজ্জ্বল ব্লব এবং দাগ সনাক্ত করতে অক্ষম ছিল, তাই আমরা এটি 0.59 এ বাড়িয়েছি।
বাইনারাইজড ইমেজটি তখন 'মিডফিল্ট 2' ফাংশন ব্যবহার করে একটি মধ্যমা ফিল্টারের মাধ্যমে প্রক্রিয়া করা হয় কারণ দ্বি-চিত্রিত ইমেজটি দ্বিমাত্রিক। ইনপুট বাইনারাইজড ইমেজে সংশ্লিষ্ট পিক্সেলের চারপাশে 5 x 5 আশেপাশের মধ্যম মান ধারণ করার জন্য আমরা প্রতিটি আউটপুট পিক্সেল সেট করি। এটি গোলমাল হ্রাস করে এবং প্রতিটি পিক্সেলের চারপাশে 5 x 5 বর্গের প্রান্ত সংরক্ষণ করে। পরবর্তীতে, আমরা 'স্ট্রেল' ব্যবহার করে একটি স্লাইডিং উইন্ডো প্রয়োগ করি, প্রতিটি ডিস্কের আশেপাশে প্রতিটি কেন্দ্রীয়, মূল পিক্সেল চিহ্নিত করার জন্য 2 এর আশেপাশের ব্যাসার্ধ সহ একটি ডিস্ক আকৃতির সমতল কাঠামো উপাদান তৈরি করতে। আমরা একটি ডিস্ক স্ট্রাকচারিং উপাদান ব্যবহার করেছি কারণ আমরা প্রতিটি বৃত্তাকার স্পট এবং প্রতিটি স্পটের মধ্যে পিক্সেল বিশ্লেষণ করছি, তাই একটি ডিস্ক আকৃতির উপাদানটি আরও দরকারী।
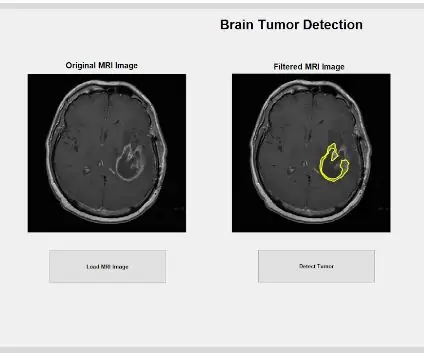
একবার ছবিটি ফিল্টার করা হয়ে গেলে, এটিকে 'ইমক্লোজ' ফাংশন ব্যবহার করে পরিষ্কার করা যেতে পারে যাতে ছবিতে ফিল্টার করা সাদা পিক্সেলের মাঝে কালো দাগ দূর করা যায় এবং এর চারপাশের সব ফাঁক বন্ধ করে দেওয়া যায়। সম্পূর্ণ প্রক্রিয়াজাত ইমেজটি পূর্ব-বরাদ্দকৃত চিত্রের দ্বিতীয় সাবপ্লটে প্লট করা যেতে পারে, যার ফলে কাঁচা এবং ফিল্টার করা ছবির মধ্যে তুলনা করা যায়।
ধাপ 4: উপবৃত্তাকার মুখোশের মাধ্যমে টিউমার বিচ্ছিন্নকরণ


টিউমারের উজ্জ্বল দাগগুলি তখন পূর্ব-উত্পন্ন উপবৃত্তাকার মুখোশের মাধ্যমে প্রধান ফিল্টার করা চিত্র থেকে বিচ্ছিন্ন করা যায়। এই মুখোশটি তৈরি করতে, আপনাকে মূল, কাঁচা এমআরআই স্ক্যান চিত্রের আকার জানতে হবে এবং এর সারি এবং কলাম দৈর্ঘ্য ব্যবহার করে যথাক্রমে x এবং y- স্থানাঙ্ক হিসাবে উপবৃত্তাকার জন্য কেন্দ্র স্থানাঙ্ক বরাদ্দ করতে হবে। আমরা y- অক্ষকে কেন্দ্র থেকে 50 ইউনিটের ব্যাসার্ধ সহ একটি প্রধান অক্ষ এবং কেন্দ্র থেকে 40 ইউনিটের ব্যাসার্ধ সহ ক্ষুদ্র অক্ষ হিসাবে সেট করি।
আমরা ম্যাটল্যাব ফাংশন 'মেশগ্রিড' ব্যবহার করে কার্টেশিয়ান প্লেন উৎপন্ন করতে দ্বি-মাত্রিক গ্রিড কো-অর্ডিনেট দিয়ে ভেক্টরের মধ্যে থাকা কো-অর্ডিনেট 1 থেকে x- অক্ষের দৈর্ঘ্য এবং 1 থেকে চিত্রের y- অক্ষের দৈর্ঘ্য । Col হল একটি ম্যাট্রিক্স যেখানে প্রতিটি সারি x- অক্ষের একটি অনুলিপি, এবং সারি হল একটি ম্যাট্রিক্স যেখানে প্রতিটি কলাম y- অক্ষের একটি অনুলিপি। কোল এবং সারির সমন্বয় দ্বারা উপস্থাপিত কার্টেশিয়ান গ্রিডের দৈর্ঘ্য (1: Y_Size) সারি এবং দৈর্ঘ্য (1: X_Size) কলাম রয়েছে। পূর্বনির্ধারিত ব্যাসার্ধ এবং কেন্দ্র স্থানাঙ্কের উপর নির্ভর করে উপবৃত্তের সমীকরণ নির্ধারণ করতে কার্টেশিয়ান গ্রিড দ্বারা উত্পন্ন কোল এবং সারির সূচকগুলি ব্যবহার করুন। উপবৃত্তাকার রূপরেখা এখন টিউমারের দাগ থেকে পাওয়া সাদা পিক্সেল দিয়ে পূর্ণ করা যায়।
প্রাক-উত্পন্ন উপবৃত্তাকার মুখোশ ব্যবহার করে আমরা ফিল্টার করা ছবি থেকে বিশ্লেষণ করতে ইচ্ছুক নির্দিষ্ট টিউমার বের করতে পারি। উপবৃত্তাকার মুখোশটি উপবৃত্তের রূপরেখার মধ্যে যৌক্তিকভাবে কোন দাগকে শনাক্ত করে এবং এটিকে টিউমার হিসেবে গ্রহণযোগ্য করার জন্য ফিল্টার করা ছবিতে একটি দাগ হিসেবে গ্রহণ করে। ফাংশন 'bwareafilt' তারপর ছবি থেকে এই সনাক্ত টিউমারের বাইরে অন্য সব বস্তু ফিল্টার করে। আমরা সমস্ত ছবির মাত্রার উপর ভিত্তি করে 500 থেকে 4000 এর একটি নির্দিষ্ট উইন্ডো ব্যবহার করেছি। আমরা তখন 'স্ট্রেল' সহ আরেকটি স্লাইডিং উইন্ডো প্রয়োগ করেছি যা ফ্ল্যাট ডিস্ক আকৃতির স্ট্রাকচারিং উপাদান হিসাবে 6 এর বৃহত্তর আশেপাশের ব্যাসার্ধের জন্য চিহ্নিত করা টিউমারের মধ্যে প্রতিটি কেন্দ্রীয় সাদা পিক্সেলের মধ্যে ফাঁক বন্ধ করে দেয়। সনাক্ত করা টিউমার স্পটটি আরও পরিষ্কার করা হয় 'ইমক্লোজ' ব্যবহার করে কালো পিক্সেলগুলিকে আরও নির্মূল করতে এবং 'ইমফিল' দিয়ে সমস্ত গর্ত পূরণ করতে। এই প্রক্রিয়াকৃত টিউমারটি বিচ্ছিন্ন টিউমার এবং এমআরআই স্ক্যানের মূল এবং ফিল্টার করা ছবিগুলির মধ্যে তুলনা করার জন্য পূর্বনির্ধারিত প্লটের তৃতীয় সাবপ্লটে প্রদর্শিত হতে পারে।
ধাপ 5: টিউমারের রূপরেখা
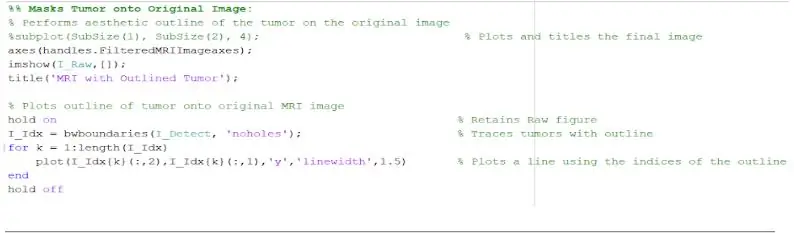
এখন যেহেতু টিউমারটি মুখোশ দিয়ে বিচ্ছিন্ন করা হয়েছে, এটির সঠিক অবস্থান দেখানোর জন্য এটি মূল চিত্রের রূপরেখা এবং প্রদর্শন করা যেতে পারে। এটি করার জন্য, আমরা একটি রূপরেখা সহ পূর্বে সনাক্ত করা টিউমারটি সনাক্ত করতে 'bwboundaries' ফাংশনটি ব্যবহার করেছি। টিউমার বস্তুর মধ্যে ছিদ্র অন্তর্ভুক্ত না করার জন্য আমরা রূপরেখা নির্দিষ্ট করেছি কারণ এটি রূপরেখা দেওয়া হচ্ছে। এটি একটি 'ফর' লুপ ব্যবহার করে মূল, কাঁচা চিত্রের উপর চক্রান্ত করা যেতে পারে যা ১.৫ পিক্সেলের প্রস্থের লাইনের সূচক ব্যবহার করে টিউমারের চারপাশে রূপরেখা তৈরি করে। এই রূপরেখাটি তখন মূল এমআরআই স্ক্যানের তুলনায় টিউমারের সঠিক আকার এবং অবস্থান দেখিয়ে কাঁচা চিত্রের উপর চক্রান্ত করা হয়।
ধাপ 6: টিউমারের শারীরিক বৈশিষ্ট্য বিশ্লেষণ করা
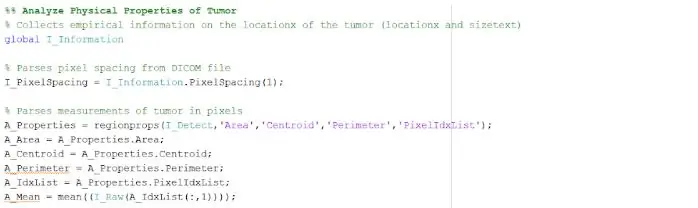
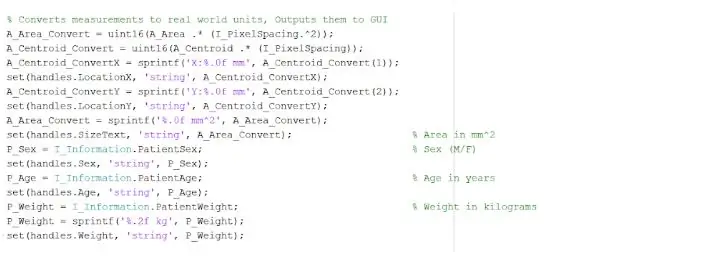
বিচ্ছিন্ন এবং রূপরেখাযুক্ত স্থানটি আমাদের টিউমারের আকার, এলাকা এবং অবস্থান সম্পর্কে দরকারী তথ্য প্রদান করতে পারে। ক্ষেত্র, পরিধি, সেন্ট্রয়েড এবং পিক্সেল সূচকের মান সম্পর্কিত টিউমারের বৈশিষ্ট্যগুলি সনাক্ত করতে আমরা 'অঞ্চলপ্রস' ফাংশনটি ব্যবহার করেছি। এই পিক্সেল ইনডেক্স মান আমাদের প্রতিটি ছবির প্রতিটি পিক্সেলের জন্য বাস্তব বিশ্ব ইউনিট দেয়, প্রতিটি স্ক্যানের জন্য অনন্য। এই বৈশিষ্ট্যগুলি তখন মিলিমিটারের বাস্তব বিশ্ব ইউনিটে রূপান্তরিত হতে পারে। প্রোগ্রামটি আমাদের যে অভিজ্ঞতাগত তথ্য প্রদান করে তা প্রতিটি এমআরআই স্ক্যানের জন্য অনন্য এবং টিউমারের আকার, অবস্থান এবং ধরন নির্ধারণে অত্যন্ত উপযোগী, যা ব্যবহারকারীরা গ্রাফিকাল ইউজার ইন্টারফেসে বিশ্লেষণ এবং অন্তর্ভুক্ত করতে পারে।
প্রস্তাবিত:
ম্যাটল্যাব ব্যবহার করে রাস্পবেরি পাই নিয়ন্ত্রণ: 5 টি ধাপ

ম্যাটল্যাব ব্যবহার করে রাস্পবেরি পাই নিয়ন্ত্রণ করা: আরে, এই টিউটোরিয়ালটি ম্যাটল্যাব ব্যবহার করে আপনার হেডলেস রাস্পবেরি পাই নিয়ন্ত্রণের বিষয়ে। নতুন রাস্পবেরি পাই বোর্ডের সমর্থনের জন্য আপনাকে সর্বশেষ সংস্করণে ম্যাটল্যাব ইনস্টল করতে হতে পারে
OpenCV ব্যবহার করে সহজ রঙ-সনাক্তকরণ: 6 টি ধাপ
OpenCV ব্যবহার করে সহজ রঙ-সনাক্তকরণ: হাই! আজ আমি ওপেনসিভি এবং পাইথন ব্যবহার করে একটি লাইভ ভিডিও থেকে একটি রঙ সনাক্ত করার একটি সহজ পদ্ধতি দেখাতে যাচ্ছি। মূলত আমি শুধু ব্যাকগ্রাউন্ড ফ্রেমে প্রয়োজনীয় রং উপস্থিত আছে কিনা তা পরীক্ষা করব এবং ওপেনসিভি মডিউল ব্যবহার করে আমি সেই অঞ্চলটি মাস্ক করব এবং
ওপেনসিভি ব্যবহার করে পাইথনে রঙ সনাক্তকরণ: 8 টি ধাপ
ওপেনসিভি ব্যবহার করে পাইথনে রঙ সনাক্তকরণ: হ্যালো! এই নির্দেশনাটি কীভাবে ওপেনসিভি লাইব্রেরি ব্যবহার করে পাইথনে একটি চিত্র থেকে একটি নির্দিষ্ট রঙ বের করতে হয় তা নির্দেশ করতে ব্যবহৃত হয়। যদি আপনি এই কৌশলটিতে নতুন হন তবে চিন্তা করবেন না, এই গাইডের শেষে আপনি আপনার নিজের রঙের প্রোগ্রাম করতে সক্ষম হবেন
রাস্পবেরি পাই এবং কণা আর্গন ব্যবহার করে কীভাবে স্মার্ট বন্যা সনাক্তকরণ অ্যালার্ম সিস্টেম তৈরি করবেন: 6 টি ধাপ

রাস্পবেরি পাই এবং পার্টিকেল আর্গন ব্যবহার করে স্মার্ট ফ্লাড ডিটেকশন অ্যালার্ম সিস্টেম কিভাবে তৈরি করবেন: আপনার বাড়ি বা কর্মস্থলে ব্যাপক ক্ষতি রোধ করার জন্য স্ট্যান্ডার্ড ফ্লাড সেন্সর থাকা খুবই ভালো। আপনি সেই স্মার্টগুলি কিনতে পারেন এই বন্যা অ্যালার্ম সিস্টেমটি কোন তরল সনাক্ত করে এবং অ্যালার ট্রিগার করে
মুখ সনাক্তকরণ এবং সনাক্তকরণ - OpenCV পাইথন এবং Arduino ব্যবহার করে Arduino ফেস আইডি: 6 ধাপ
মুখ সনাক্তকরণ এবং সনাক্তকরণ | ওপেনসিভি পাইথন এবং আরডুইনো ব্যবহার করে আরডুইনো ফেস আইডি: মুখের স্বীকৃতি AKA ফেস আইডি আজকাল মোবাইল ফোনের অন্যতম গুরুত্বপূর্ণ বৈশিষ্ট্য। সুতরাং, আমার একটি প্রশ্ন ছিল " আমি কি আমার আরডুইনো প্রকল্পের জন্য একটি ফেস আইডি রাখতে পারি " এবং উত্তর হল হ্যাঁ … আমার যাত্রা নিম্নরূপ শুরু হয়েছিল: ধাপ 1: আমাদের প্রবেশাধিকার
